1. リボソーム改変による大腸菌の宿主開発
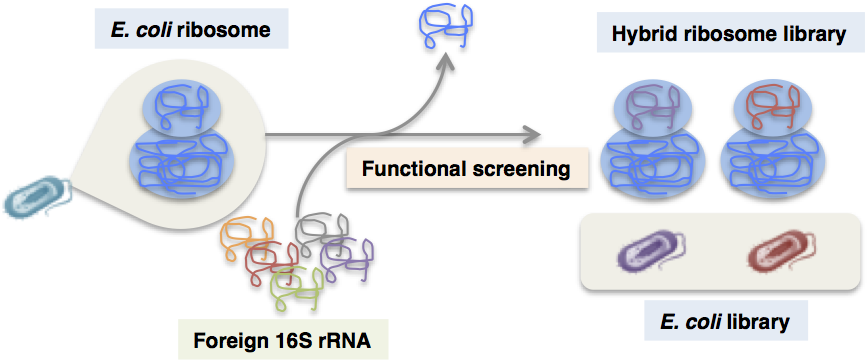
大腸菌は、組換えDNA実験から物質生産まで、最も汎用的に使われる宿主微生物です。我々は、細胞内の翻訳装置であるリボソームを改変することにより、大腸菌の諸性質(表現型)を様々に変化させることに成功しました。本技術を有用宿主開発に活かすための技術開発を進めています。
主な発表論文
- Miyazaki K, Kitahara K (2018) Functional metagenomic approach to identify overlooked antibiotic resistance mutations in bacterial rRNA. Sci Rep 8:5179
- Tsukuda M, Kitahara K, Miyazaki K (2017) Comparative RNA function analysis reveals high functional similarity between distantly related bacterial 16S rRNAs. Sci Rep 7:9993
- Sato M, Miyazaki K (2017) Phylogenetic network analysis revealed the occurrence of horizontal gene transfer of 16S rRNA in the genus Enterobacter. Front Microbiol 8:2225
- Kitahara K, Yasutake Y, Miyazaki K (2012) Mutational robustness of 16S ribosomal RNA, shown by experimental horizontal gene transfer in Escherichia coli. Proc Natl Acad Sci USA 109(47):19220-19225
- Kitahara K, Miyazaki K (2011) Specific inhibition of bacterial RNase T2 by helix 41 of 16S ribosomal RNA. Nat Commun 2:549
2. メタゲノミクスを活用した有用酵素の探索
環境中に生育する細菌の99%は、実験室での分離培養が困難であると言われています。そのため、分離培養に基礎をおく従来の手法では、環境微生物の実態が明らかにされず、その利用も限定的でした。これに対し我々は、培養行程を経ずに環境試料から分離した様々なバクテリア由来の混じりもののゲノムを解析する〈メタゲノム技術〉により難培養性の問題の解決を図ります。有用遺伝子資源の宝庫として注目されるメタゲノムの高度利用、それを可能にするスクリーニング技術の開発などを行っています。
主な発表論文
- Uchiyama T, Yaoi K and Miyazaki K (2015) Glucose-tolerant β-glucosidase retrieved from a Kusaya gravy metagenome. Front Microbiol 6:548
- Uchiyama T, Miyazaki K (2013) Metagenomic screening for aromatic compound-responsive transcriptional regulators. PLOS ONE 8(9):e75795
- Uchiyama T, Miyazaki K, Yaoi K (2013) Characterization of a novel β-glucosidase from a compost microbial metagenome with strong transglycosylation activity. J Biol Chem 288(25):18325-18334
- Verma D, Kawarabayasi Y, Miyazaki K, Satyanarayana T (2013) Cloning, expression and characteristics of a novel alkalistable and thermostable xylanase encoding gene (mxyl) retrieved from compost-soil metagenome. PLOS ONE 8(1):e52459
- Uchiyama T, Miyazaki K (2010) Product-induced gene expression, a product-responsive reporter assay used to screen metagenomic libraries for enzyme-encoding genes. Appl Environ Microbiol 76(21):7029-7035
- Suenaga H, Koyama Y, Miyakoshi M, Miyazaki R, Yano H, Sota M, Ohtsubo Y, Tsuda, M, Miyazaki K (2009) Novel organization of aromatic degradation pathway genes in a microbial community as revealed by metagenomic analysis. ISME J 3(12):1335-1348
- Suenaga H, Ohnuki T, Miyazaki K (2007) Functional screening of a metagenomic library for genes involved in microbial degradation of aromatic compounds. Environ Microbiol 9(9):2289-2297
3. 進化分子工学による生体分子の機能改変
いかに精密かつ巧妙に機能する蛋白質、あるいはさらに高次な生物機能も、それらは例外なく進化 ~多様性の創出と選択~ の所産であり、何ら知的な造作物ではありません。生物が望むと望まざると身につけている〈進化〉という属性が、高度に洗練された生物機能を実現します。〈進化〉は、膨大な試行錯誤の産物であるため、一見非合理的な方法であると考えられていますが、進化は成功に裏付けられた万能なものづくり技術なのです。人類はこれまでにも品種改良・育種を通して〈進化〉を利用してきました。我々は〈進化によるものづくり ~進化分子工学~〉を蛋白質、遺伝子、酵素に適用し、高機能化すべく、加速進化の方法論、遺伝子変異手法やライブラリ構築技術の開発、応用研究まで幅広く行っています。
主な発表論文
- Miyazaki K (2018) Molecular engineering of the salicylate-inducible transcription factor Sal7AR for orthogonal and high gene expression in Escherichia coli. PLoS One 13(4):e0194090
- Tsukuda M, Miyazaki K (2013) Directed evolution study unveiling key sequence factors that affect translation efficiency in Escherichia coli. J Biosci Bioeng 116(5):540-545.
- Miyazaki K, Takenouchi M, Kondo H, Noro N, Suzuki M, Tsuda S (2006) Thermal stabilization of Bacillus subtilis family-11 xylanase by directed evolution. J Biol Chem 281(15):10236-10242
- Miyazaki K, Takenouchi M (2002) Creating random mutagenesis libraries using megaprimer PCR of whole plasmid. Biotechniques 33(5):1033-1034, 1036-1038
- Miyazaki K, Wintrode PL, Grayling RA, Rubingh DN, Arnold FH (2000) Directed evolution study of temperature adaptation in a psychrophilic enzyme. J Mol Biol 297(4):1015-1026
- Miyazaki K, Arnold FH (1999) Exploring nonnatural evolutionary pathways by saturation mutagenesis: rapid improvement of protein function. J Mol Evol 49(6):716-720