タンパク質の糖鎖修飾を「見える化」するソフトウエア「GRable Version 1.0」-創薬に有用な糖タンパク質の探索・評価を加速-
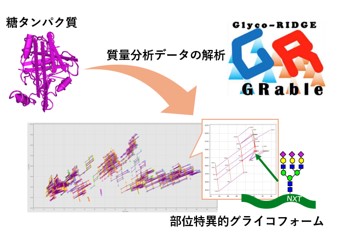
細胞分子工学研究部門 分子細胞マルチオミクス研究グループの岡谷千晶 主任研究員、富永大介 主任研究員(当時、現:明治薬科大学教授)、富岡あづさ テクニカルスタッフ、坂上弘明 研究員、久野敦 研究グループ長と、名古屋大学 糖鎖生命コア研究所糖鎖ビッグデータセンターの梶裕之 特任教授(兼:産総研 客員研究員)、慶應義塾大学 医学部の洪繁 特任准教授、合田徳夫 特任講師(当時)は、糖鎖が結合したタンパク質の質量分析によるグライコプロテオーム解析データを自動解析できるソフトウエア「GRable Version 1.0(以下、GRable)」を開発しました。
糖ペプチドの同定は2段階の質量分析(MS2)による方法が主流ですが、産総研では、1段階の分析(MS1)で糖ペプチドシグナルを特定する方法(Glycan heterogeneity-based Relational IDentification of Glycopeptide signals on Elution profile(Glyco-RIDGE)法)を開発し、MS2による方法よりも網羅性の高い分析を可能としました。本ソフトウエアはGlyco-RIDGE法に基づく質量分析を支援するもので、バイオ医薬品などの特定糖タンパク質の詳細構造解析や、創薬シーズとなり得る糖タンパク質探索のための大規模解析のどちらにも活用できることを実証しました。本ソフトウエアはウェブ公開されており、無償で利用可能です[1-3]。
なお、この技術の詳細は、2024年9月30日に「Molecular & Cellular Proteomics」に掲載されました。
※詳細は図下のプレスリリースをご覧ください。
プレスリリース
投稿論文
- 論文タイトル:GRable version 1.0: A software tool for site-specific glycoform analysis with improved MS1-based glycopeptide detection with parallel clustering and confidence evaluation with MS2 information
- 著者:Chiaki Nagai-Okatani*, Daisuke Tominaga, Azusa Tomioka, Hiroaki Sakaue, Norio Goda, Shigeru Ko, Atsushi Kuno, Hiroyuki Kaji*(*:責任著者)
- 雑誌:Molecular & Cellular Proteomics
関連ウェブサイト
1.GRableウェブサイト
2.GRable紹介ページ(細胞分子工学研究部門:公開ツール)
https://unit.aist.go.jp/cmb5/ja/result/tool.html/
3.質量分析によるグライコプロテオーム解析を加速するソフトウェア「GRable」の公開(細胞分子工学部門:トピックス)