細胞動態システム研究グループ
研究概要
成長が期待されているバイオ医薬品、再生医療、細胞食品といった細胞産業では、従来の製造プロセスとは異なる細胞制御が必要となる。 細胞産業における製品開発や製造プロセスを最適化するために、細胞や動物の動態変化解析とオミクス情報解析を集約させ、細胞の操作や改変、品質管理に必要な新規技術などを開発する。
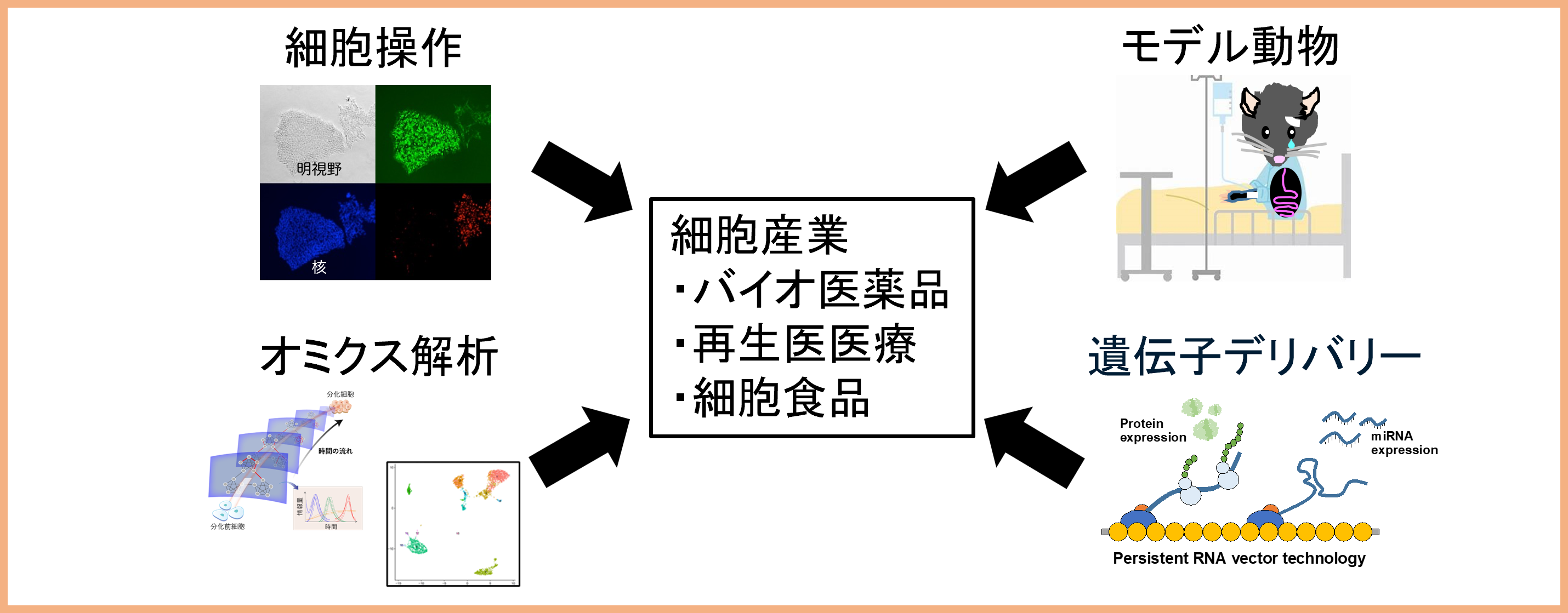
研究課題
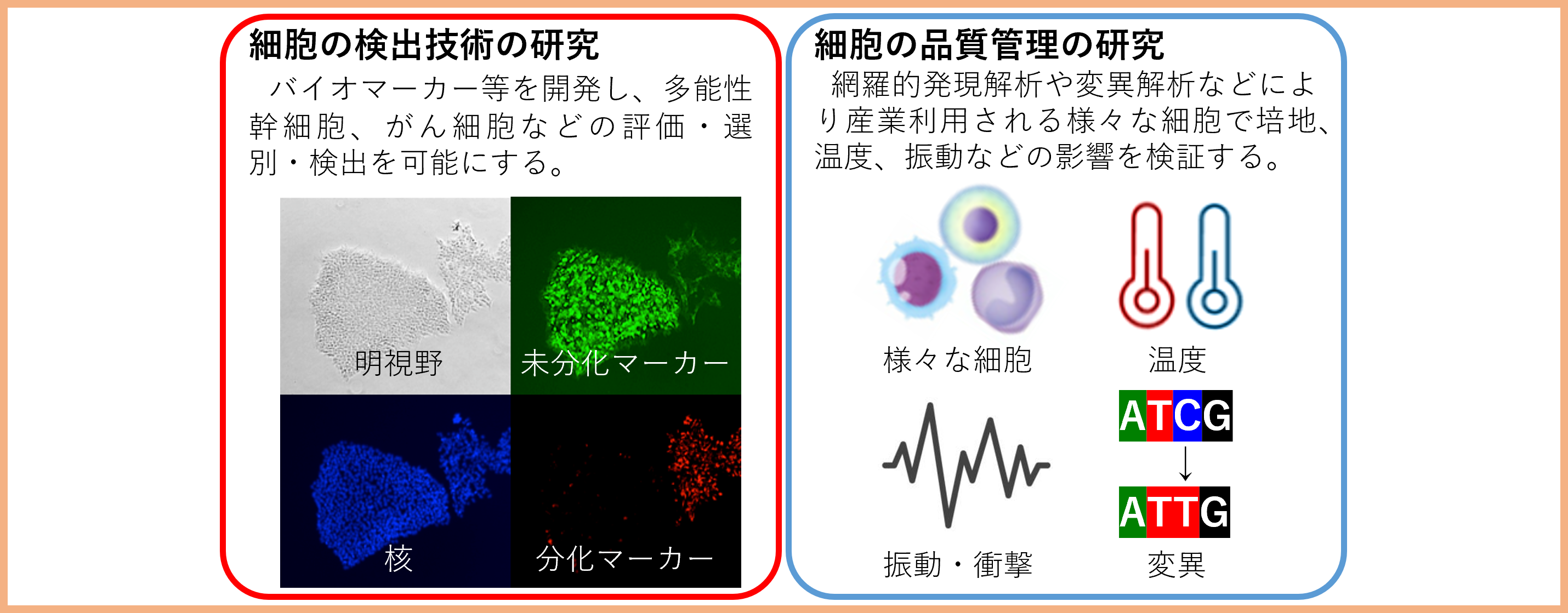
研究課題1:産業用細胞の操作・管理技術の開発
研究担当者:回渕 修治
再生医療、細胞性食品などの細胞を用いた産業における製造プロセスでは、生きた細胞を扱うために既存の産業とは異なる操作・管理技術が必要となる。我々はこれまで、様々な細胞の検出技術や品質管理の研究を進め、企業との共同研究や技術の実用化を行ってきました。今後も新しい技術の開発を目指します。
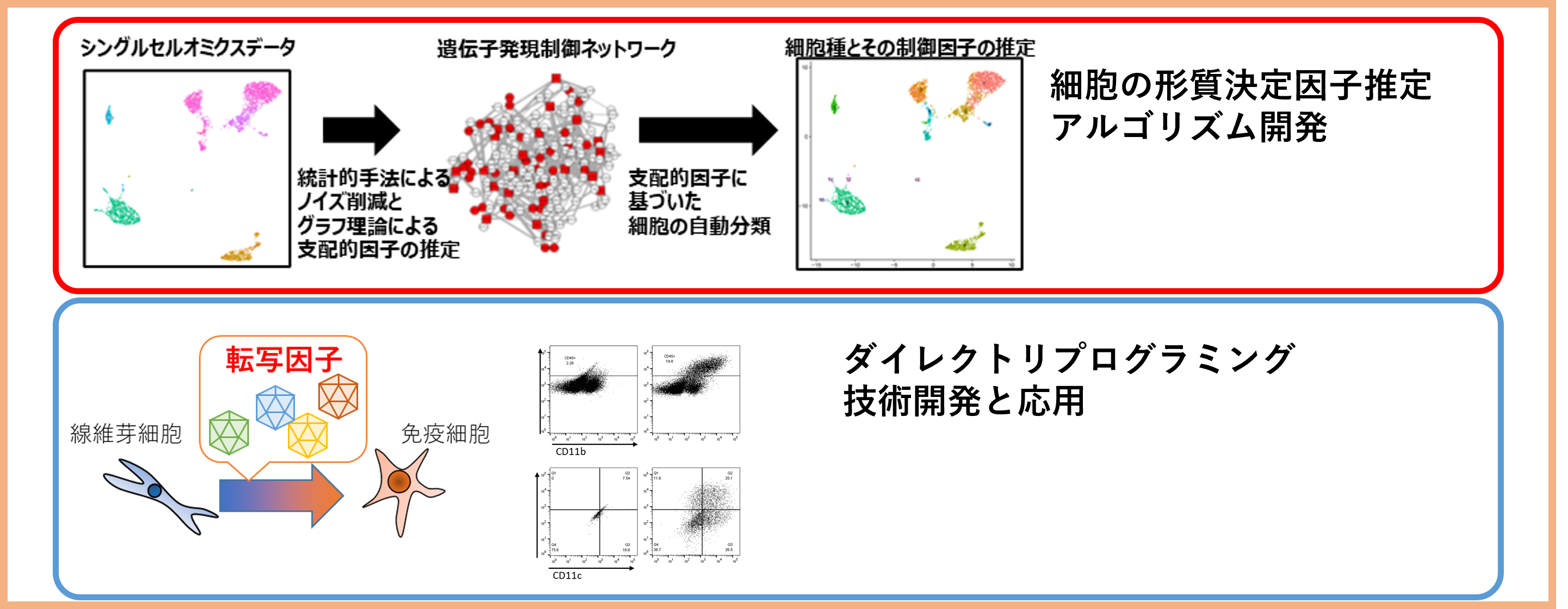
研究課題2:細胞形質の決定因子を推定するアルゴリズムの開発と応用
研究担当者:熊谷 雄太郎
医療、食品などに用いる細胞の製造・開発において目的の形質を持つ細胞を系統的に作り出すことが求められます。我々はオミクス解析(メタボローム、プロテオーム、トランスクリプトームなど)から得られるマルチモーダルデータを統合解析し、細胞の形質を決定する因子を推定するアルゴリズムを開発し、それを応用することで新規の形質を持つ細胞を作製、利用することを目指しています。また、細胞間・臓器間の相互作用を記述しシミュレートする技術への応用も目指します。
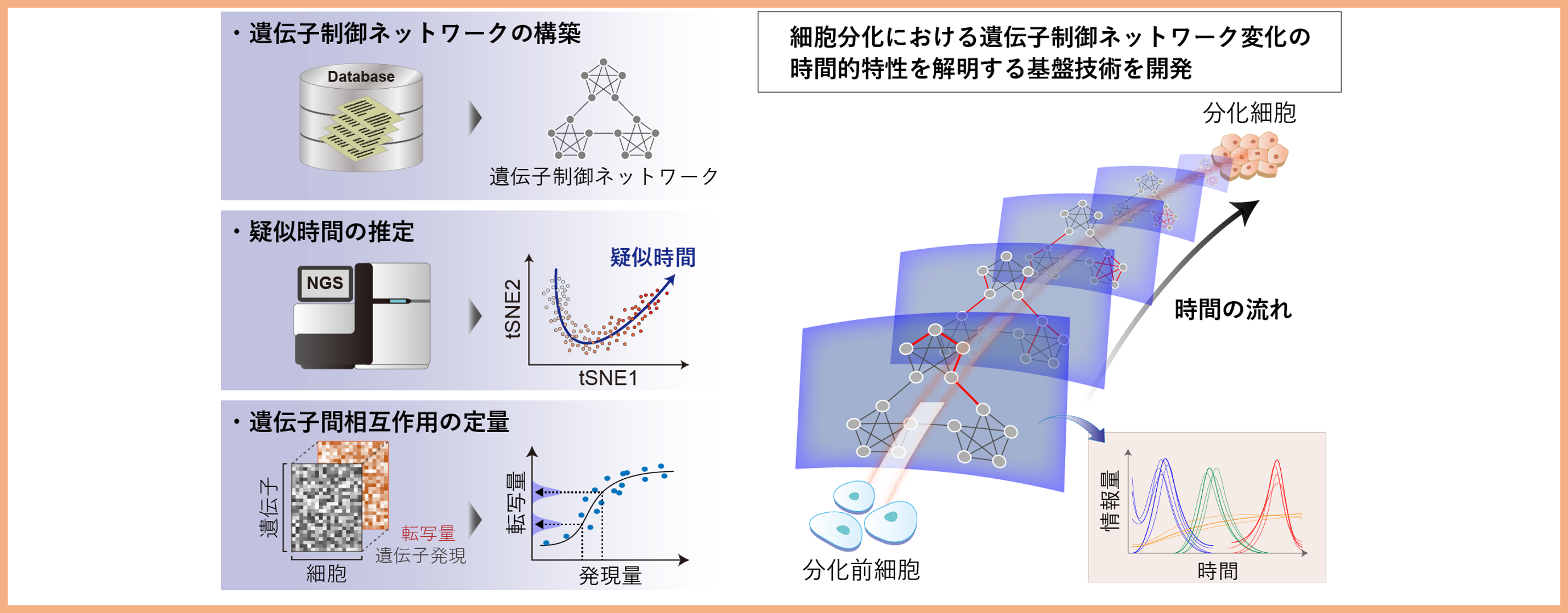
研究課題3:細胞内制御ネットワークの動的特性を解析する基盤技術の構築
研究担当者:川田 健太郎
細胞内には多種多様な分子が存在し、これらが互いに情報をやり取りすることで細胞の状態が決定されます。本研究では、細胞内分子が情報を伝えるネットワークが、細胞の分化や外部環境への適応に伴ってどのように変化するのか、ネットワークの変化が細胞の状態にどのように影響を与えるかを高精度で解析する基盤技術を開発しています。また、これらの研究を通じて多能性幹細胞などにおける分化制御の高精度化を推進しています。
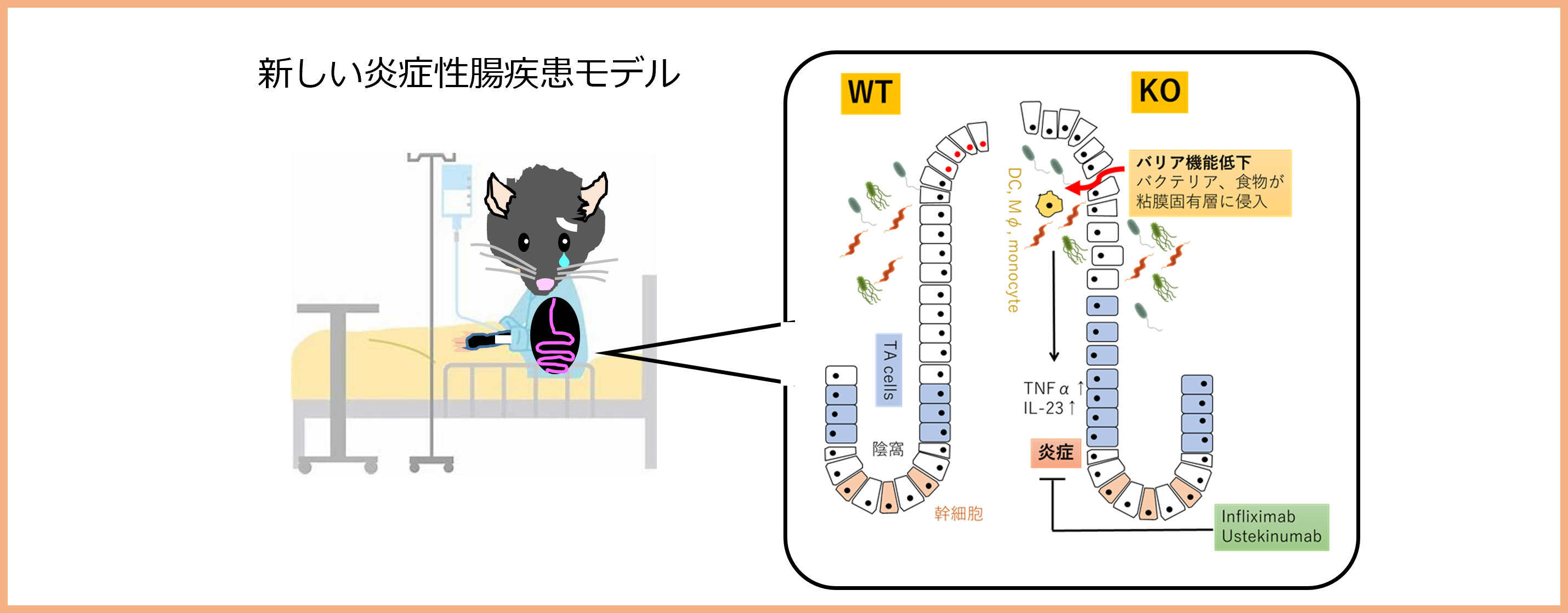
研究課題4:健康長寿社会の実現:慢性炎症性疾患の発症機構解明
研究担当者:佐々木 保典
私たちはあるタンパク質を研究対象としています。このタンパク質は多種多様な生命現象・疾患、特にストレス応答に関係し、生体にリジリエンスを与えることがわかってきました。このタンパク質遺伝子を機能破壊したマウスでは、消化器系のバリア機能低下の他、慢性炎症性疾患様の症状が認められました。そこで、このマウスを炎症性腸疾患をはじめとする慢性炎症性疾患の発症機構の解明に活用していきます。
グループの構成メンバー
| 顔写真 | 所属・役職および名前 | 専門分野 | その他、etc |
|---|---|---|---|
 |
グループ長 回渕 修治 |
|
|
 |
主任研究員 佐々木 保典 |
|
|
 |
主任研究員 熊谷 雄太郎 |
|
|
 |
研究グループ付(兼務) 川田 健太郎 |
|
業績リスト
- Zhang, Y; Yang, W; Kumagai, Y; Loza, M; Yang, Y; Park, SJ; Nakai, K.
In Silico Analysis Revealed Marco (SR-A6) and Abca1/2 as Potential Regulators of Lipid Metabolism in M1 Macrophage Hysteresis.
INT J MOL SCI. 2024 Dec 26. doi:10.3390/ijms26010111 - Kusano, T; Sotani, Y; Takeda, R; Hatano, A; Kawata, K; Kano, R; Matsumoto, M; Kano, Y; Hoshino, D.
Time-series transcriptomics reveals distinctive mRNA expression dynamics associated with gene ontology specificity and protein expression in skeletal muscle after electrical stimulation-induced resistance exercise.
FASEB J. 2024 Nov 30;38(22):e70153. doi:10.1096/fj.202401420RR - Zhou, J; Sekiguchi, Y; Sano, M; Nishimura, K; Hisatake, K; Fukuda, A.
A Sendai virus-based expression system directs efficient induction of chondrocytes by transcription factor-mediated reprogramming.
SCI REP. 2024 Oct 29;14(1):26004. doi: 10.1038/s41598-024-77508-1 - Mawaribuchi, S; Iida, M; Haramoto, Y.
Fusion of breast cancer MCF-7 cells with mesenchymal stem cells rearranges interallelic gene expression and enhances cancer malignancy.
BIOCHEM BIOPHYS RES COMMUN. 2024 Oct 23:736:150887. doi: 10.1016/j.bbrc.2024.150887 - Kumagai, Y.
BootCellNet, a resampling-based procedure, promotes unsupervised identification of cell populations via robust inference of gene regulatory networks.
PLOS COMPUT BIOL. 2024 Sep 30;20(9):e1012480. doi: 10.1371/journal.pcbi.1012480 - Miura, I; Shams, F; Ohki, J; Tagami, M; Fujita, H; Kuwana, C; Nanba, C; Matsuo, T Ogata, M; Mawaribuchi, S; Shimizu, N; Ezaz, T.
Multiple Transitions between Y Chromosome and Autosome in Tagos Brown Frog Species Complex.
GENES (Basel). 2024 Feb 26;15(3):300. doi: 10.3390/genes15030300 - Kishimoto, T; Nishimura, K; Morishita, K; Fukuda, A; Miyamae, Y; Kumagai, Y; Sumaru, K; Nakanishi, M; Hisatake, K; Sano, M.
An engineered ligand-responsive Csy4 endoribonuclease controls transgene expression from Sendai virus vectors.
J BIOL ENG. 2024 Jan 16;18(1):9. doi: 10.1186/s13036-024-00404-9 - Zhang, Y; Yang, W; Kumagai, Y; Loza, M; Zhang, W; Park, SJ; Nakai, K.
Multi-omics computational analysis unveils the involvement of AP-1 and CTCF in hysteresis of chromatin states during macrophage polarization.
FRONT IMMUNOL. 2023 Dec 20;14:1304778. doi: 10.3389/fimmu.2023.1304778 - Takeda, N; Tsuchiya, A; Mito, M; Natsui, K; Natusi, Y; Koseki, Y; Tomiyoshi, K; Yamazaki, F; Yoshida, Y; Abe, H; Sano, M; Kido, T; Yoshioka, Y; Kikuta, J; Itoh, T; Nishimura, K; Ishii, M; Ochiya, T; Miyajima, A; Terai, S.
Analysis of distribution, collection, and confirmation of capacity dependency of small extracellular vesicles toward a therapy for liver cirrhosis.
INFLAMM REGEN. 2023 Oct 9;43(1):48. doi: 10.1186/s41232-023-00299-x - Kumagai, Y; Saito, Y; Kida, YS.
A multiomics atlas of brown adipose tissue development over time.
ENDOCRINOLOGY. 2023 Apr 21:bqad064. doi: 10.1210/endocr/bqad064 - Mawaribuchi, S; Ito, M; Ogata, M; Yoshimura, Y; Miura, I.
Parallel Evolution of Sex-Linked Genes across XX/XY and ZZ/ZW Sex Chromosome Systems in the Frog Glandirana rugosa.
GENES (Basel). 2023 Jan 18;14(2):257. doi: 10.3390/genes14020257 - Mawaribuchi, S; Shimomura, O; Oda, T; Hiemori, K; Shimizu, K; Yamase, K; Date, M; Tateno, H.
rBC2LCN-reactive SERPINA3 is a glycobiomarker candidate for pancreatic ductal adenocarcinoma.
GLYCOBIOLOGY. 2023 Feb 2:cwad009. doi: 10.1093/glycob/cwad009 - Huang, T; Sato, Y; Kuramochi, A; Ohba, Y; Sano, M; Miyagishi, M; Tateno, H; Wadhwa, R; Kawasaki, K; Uchida, T; Ekdahl, KN; Nilsson, B; Chung, UI; Teramura, Y.
Surface modulation of extracellular vesicles with cell-penetrating peptide-conjugated lipids for improvement of intracellular delivery to endothelial cells.
REGEN THER. 2023 Jan 11;22:90-98. doi: 10.1016/j.reth.2022.12.007 - Mawaribuchi, S; Haramoto, Y; Ikeda, N; Ito, M.
Evolutionary features of ligands and their receptors via protein-protein interactions and essentiality in primates.
GENES CELLS. 2023 Jan 9. doi: 10.1111/gtc.13006 - Imai, Y;Mori, N; Nihashi, Y; Kumagai, Y; Shibuya, Y; Oshima, J; Sasaki, M; Sasaki, K; Aihara, Y; Sekido, M; Kida, YS.
Therapeutic Potential of Adipose Stem Cell-Derived Conditioned Medium on Scar Contraction Model.
BIOMEDICINES. 2022 Sep 24;10(10):2388. doi: 10.3390/biomedicines10102388 - Sano, M; Morishita, K; Onizawa, Y; Takagi, T; Sumaru, K.
Rapid and Highly Sensitive Method for Evaluating Surface Coatings against an Enveloped RNA Virus.
ACS APPL BIO MATER. 2022 Oct 14. doi: 10.1021/acsabm.2c00613 - Katsumi, T; Shams, F; Yanagi, H; Ohnishi, T; Toda, M; Lin, SM; Mawaribuchi, S; Shimizu, N; Ezaz, T; Miura, I.
Highly rapid and diverse sex chromosome evolution in the Odorrana frog species complex.
DEV GROWTH DIFFER. 2022 Jul 26. doi: 10.1111/dgd.12800 - Omoto, T; Yimiti, D; Sanada, Y; Toriyama, M; Ding, C; Hayashi, Y; Ikuta, Y; Nakasa, T; Ishikawa, M; Sano, M; Lee, M; Akimoto, T; Shukunami, C; Miyaki, S; Adachi, N.
Tendon-Specific Dicer Deficient Mice Exhibit Hypoplastic Tendon Through the Downregulation of Tendon-Related Genes and MicroRNAs.
FRONT CELL DEV BIOL. 2022 Jun 14;10:898428. doi: 10.3389/fcell.2022.898428 - Miura, I; Shams, F; Jeffries, DL; Katsura, Y; Mawaribuchi, S; Perrin, N; Ito, M; Ogata, M; Ezaz, T.
Identification of ancestral sex chromosomes in the frog Glandirana rugosa bearing XX-XY and ZZ-ZW sex-determining systems.
MOL ECOL. 2022 Jun 12. doi: 10.1111/mec.16551 - Sugisawa, E; Kondo, T; Kumagai, Y; Kato, H; Takayama, Y; Isohashi, K; Shimosegawa, E; Takemura, N; Hayashi, Y; Sasaki, T; Martino, MM; Tominaga, M; Maruyama, K.
Nociceptor-derived Reg3γ prevents endotoxic death by targeting kynurenine pathway in microglia.
CELL REP. 2022 Mar 8;38(10):110462. doi: 10.1016/j.celrep.2022.110462 - Sano, M; Morishita, K; Oikawa, S; Akimoto, T; Sumaru, K; Kato, Y.
Live-cell imaging of microRNA expression with post-transcriptional feedback control.
MOL THER NUCLEIC ACIDS. 2021 Aug 26;26:547-556. doi: 10.1016/j.omtn.2021.08.018 - Ogita, Y; Tamura, K; Mawaribuchi, S; Takamatsu, N; Ito, M.
Independent pseudogenizations and losses of sox15 during amniote diversification following asymmetric ohnolog evolution.
BMC ECOL EVOL. 2021 Jun 30;21(1):134. doi: 10.1186/s12862-021-01864-z